本文为原创文章,未经本人允许,禁止转载。转载请注明出处。
1.Introduction
体数据是生物医学数据中非常常见的一种形式。由于计算机屏幕一次只能显示一张2D切片,所以体数据分割标签的标注非常麻烦。一张切片接一张切片的去标注是非常枯燥乏味的。并且这样做效率很低,因为相邻两张切片非常近似。特别是对于需要大量标注数据的方法来说,创建一个拥有完整标注的3D数据的数据集并不是一个有效的方法。
在本文中,我们提出了一种深度网络,其训练只需要部分标注的2D切片,即可生成dense volumetric segmentation。该网络可以以两种不同的方式使用,如Fig1所示:第一种方式是从稀疏标注的数据集中生成dense segmentation;第二种方式是从多个稀疏标注的数据集中学习,从而推广到新数据上。这两种方式高度相关。
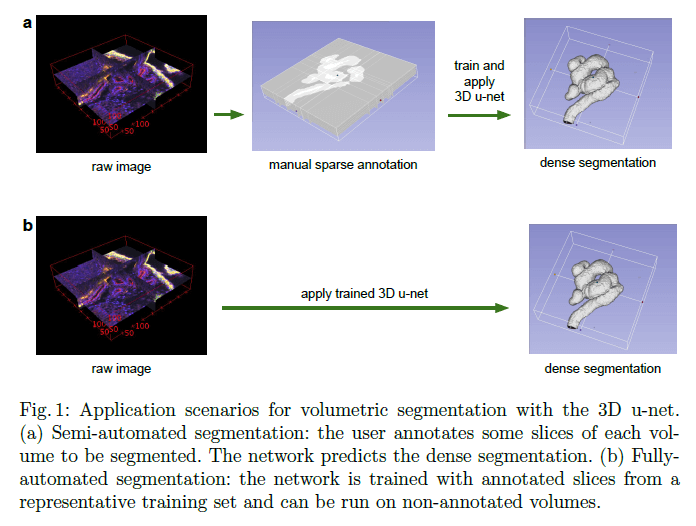
Fig1展示了3D u-net的应用场景。(a)半自动分割:用户只需标注每个volume中的部分slice。网络便可预测出dense segmentation。(b)全自动分割:网络在带有标注slice的训练集上进行训练,然后运行在无标注的volume上。
该网络基于之前的u-net框架。虽然u-net是一个2D框架,但本文提出的网络以3D体数据作为输入,并通过对应的3D操作来处理这些数据,比如3D卷积、3D max pooling和3D上卷积层(up-convolutional layers)。此外,我们避免了网络框架中的瓶颈结构,并使用BatchNorm来加快收敛。
对于许多生物医学应用,只需要很少的图像就可以训练出一个泛化良好的网络。这是因为每张图像都包括有相应变化的重复结构。对于体数据,这种影响更加明显,因此我们可以在两组体数据上训练网络,然后再推广到第三组体数据上。加权损失函数和特殊的数据扩展方式使得我们可以用很少的手动标注slice(即稀疏标注的训练数据)来训练网络。
我们所提出的方法在一些非常困难的数据集上也取得了不错的表现。此外,我们还进行了定性评估和定量评估。并且试验了标注的slice数量对网络性能的影响。使用Caffe实现网络,开源地址:http://lmb.informatik.uni-freiburg.de/resources/opensource/unet.en.html。
1.1.Related Work
如今,对于具有挑战性的2D医学图像分割任务,CNN表现已经接近人类。鉴于此,目前有很多研究尝试将3D CNN网络用于生物医学体数据。
2.Network Architecture
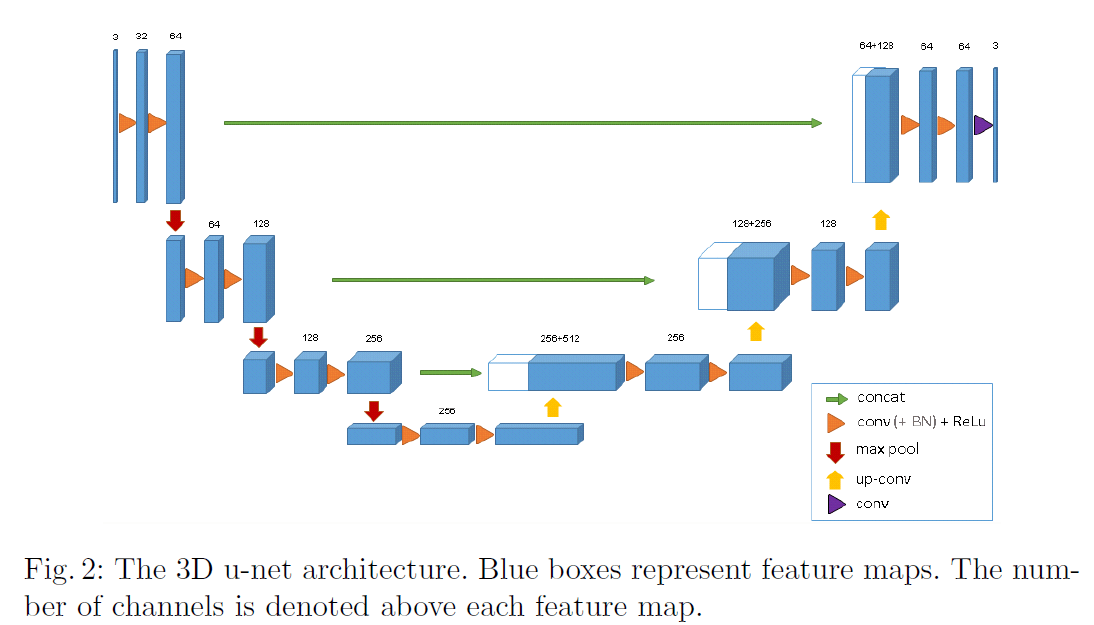
Fig2是3D u-net的网络框架。整体类似于u-net,分为analysis path和synthesis path,都有4个resolution step(个人理解:其实UNet是4步,3D u-net是3步)。网络的输入大小为$132 \times 132 \times 116$的3通道数据,网络中的卷积都是$3\times 3 \times 3$的多通道3D卷积(除了最后一层),max-pooling所用的核大小都是$2 \times 2 \times 2$(在3个方向上步长都是2),卷积后的激活函数都为ReLU,上采样所用的核大小也都是$2 \times 2 \times 2$(在3个方向上步长都是2)。最后一层的卷积核大小为$1 \times 1 \times 1$,输出通道数为3,对应3个label。网络一共有19069955个参数。我们通过在max-pooling之前将通道数翻倍来避免瓶颈结构。我们在synthesis path中也使用这种策略来避免瓶颈结构。
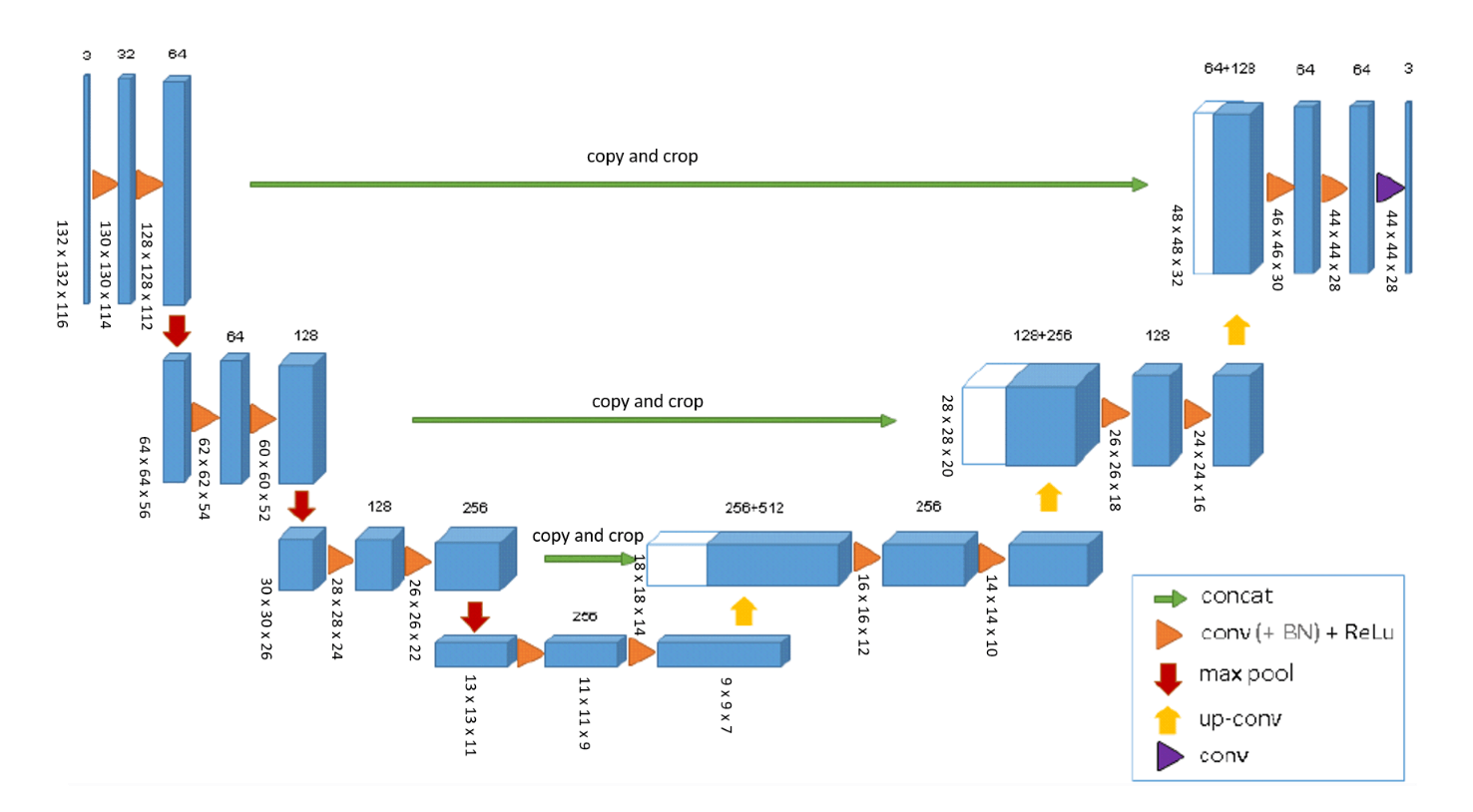
如上图所示,网络的输入为$132 \times 132 \times 116$的3通道数据。输出大小为$44 \times 44 \times 28$。一个体素的大小是$1.76 \times 1.76 \times 2.04 \mu m^3$,在分割结果中,每个体素的感受野近似是$155 \times 155 \times 180 \mu m^3$。因此,每个输出体素都有足够的上下文来进行有效的学习。
在ReLU之前还使用了BN。因为我们的batch size只有几个样本,甚至只有1个样本。所以BN中所用的均值和方差来自训练阶段,这样效果会比较好。
我们可以在稀疏注释的数据上进行训练的关键在于使用了加权的softmax损失函数。将未标注像素点的权重设为0,这样使得我们可以只从有标注像素点上进行学习,这一策略可以扩展到整个volume。
3.Implementation Details
3.1.Data
我们有三个Xenopus kidney embryos的样本,都位于Nieuwkoop-Faber stage 36-37阶段。其中一个见Fig1左图。数据的介绍比较专业化,本博文省去该部分,有兴趣的可以直接点击博文末尾的原文链接去阅读原文。
3.2.Training
采用了一些诸如旋转、缩放等数据增强的办法。在NVIDIA TitanX GPU上训练了70000次迭代,共耗时3天。
4.Experiments
4.1.Semi-Automated Segmentation
半自动分割的示例见Fig3。

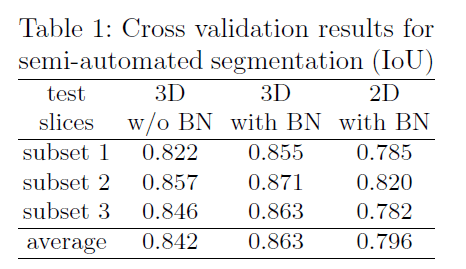
3D和2D模型的比较结果见表1,2D模型将每个slice视为独立的图像。
我们还分析了带标注的slice的数量对网络性能的影响。测试结果见表2。
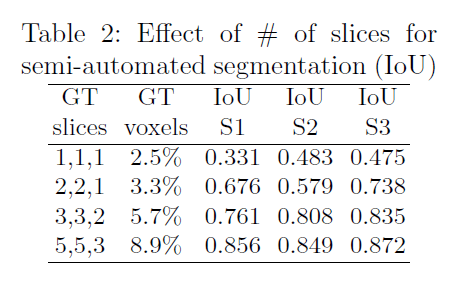
“GT slices”列指的是在3个正交方向上带有标注的slice的数量。S1、S2、S3是3个不同的测试样本。
4.2.Fully-automated Segmentation
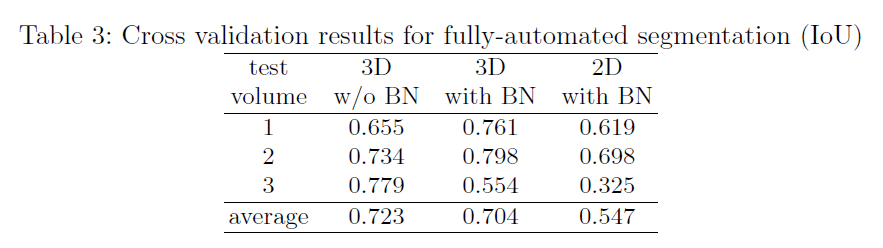
和2D模型的比较见表3。和半自动分割的结果不同,对于3D模型,BN反倒降低了性能,我们认为数据集的巨大差异是造成这个问题的原因。
5.Conclusion
3D unet是一个端到端的方法,支持半自动分割和自动分割。文中的网络都是train from scratch,也没有进行优化。该模型适用于医学3D数据的分割任务。
6.原文链接
👽3D U-Net:Learning Dense Volumetric Segmentation from Sparse Annotation