本文为原创文章,未经本人允许,禁止转载。转载请注明出处。
1.Introduction
CNN目前是医学图像分割领域内的主流方法。然而,每个分割的benchmark似乎都需要专门的框架和特定的训练策略才能达到有竞争力的性能表现。这导致很多方法仅在一个或少数几个数据集上验证过,其很难在有限的场景之外达到承诺的性能表现。医学分割十项全能(Medical Segmentation Decathlon)旨在解决这个问题:这项挑战要求参与者创建一种分割算法,其需要在与人体不同实体相对应的10个数据集上进行泛化测试。这些算法可以动态地适应特定数据集的特性,但仅允许以完全自动的方式这样做。挑战分为两个连续的阶段:1)开发阶段。即参与者可以使用7个数据集来优化他们的方法,并提交最终模型(不再改变)以及在对应7个测试集上的分割结果。2)第二个阶段是在另外3个未公开的数据集上评估已提交的模型。
虽然U-Net通常被用作这类任务的benchmark,但由于特定的框架结构、预处理、训练、推理以及后处理之间的相互依赖,通常导致U-Net作为benchmark时表现不佳。此外,如果网络尚未针对手头的任务进行完全优化,那么一些用于提高网络性能的调整可以很容易的奏效。但是,在我们自己的初步实验中,这些调整无法在已经完全优化的网络中改善分割结果,因此也很可能无法提高现有技术的水平。这让我们相信非网络框架的部分在分割任务中的影响更大,其作用被严重低估了。
本文我们提出了nnU-Net(“no-new-Net”)框架。其基于三个简单的U-Net模型,这三个模型只是对原始U-Net进行了微小的修改。我们没有使用近期提出的一些改进策略,比如残差连接、密集连接或注意力机制等。nnU-Net会根据给定图像的几何结构自动调整其框架。更重要的是,nnU-Net彻底定义了以下所有步骤。这些步骤会极大的影响网络性能:预处理(比如重采样和归一化)、训练(比如loss和优化器的设置,数据扩展等)、推理(比如基于patch的策略,模型集成)和潜在的后处理(比如强制使用单个连通域)。
2.Methods
2.1.Network architectures
医学图像通常有三个维度,这就是为什么我们考虑将一个2D U-Net、一个3D U-Net和一个级联U-Net作为我们基础的U-Net框架。2D U-Net和3D U-Net用来产生全分辨率的分割,级联则首先产生低分辨率的分割,然后再逐步refine。和原始的U-Net相比,我们的修改几乎可以忽略不计,相反,我们将精力集中在设计自动化的训练pipeline。
U-Net是一个成功的encoder-decoder网络,近年来受到了很多关注。其encoder部分的工作原理类似于传统的CNN分类网络,以减少空间信息为代价,连续地聚合语义信息。由于在分割任务中,语义和空间信息对网络的成功至关重要,因此必须以某种方式恢复丢失的空间信息。U-Net通过decoder来实现这一点。和其他分割网络不同,比如FCN和DeepLab,U-Net可以更好的分割精细结构。
我们和原始U-Net不一样的地方在于:1)使用了Leaky ReLU(常数为$1e^{-2}$)而没有使用ReLU;2)使用了instance normalization而不是batch norm。
instance normalization(实例归一化)是一种常用的深度学习中的归一化技术,它通过对每个样本的每个通道进行独立的标准化来规范化网络层的输出。在计算机视觉任务中,例如图像分割和图像生成等领域,实例归一化已被证明是一种有效的正则化技术,可以提高网络的泛化能力和鲁棒性。
实例归一化的计算方法是将每个样本在通道维度上做归一化,即先计算每个样本在每个通道上的均值和标准差,然后对每个样本的每个通道进行独立的标准化,得到规范化后的输出。
从直观来说,使用2D U-Net来分割3D医学图像是一个次优的选择,因为z方向的信息不能被很好的利用。然而,有证据证明如果数据集是各向异性(anisotropic)的,那么传统的3D分割方法性能反而不好(可参见十项全能挑战中的前列腺数据集)。
在图像处理和计算机视觉中,各向同性(isotropic)是指在各个方向上具有相同的特性,而各向异性(anisotropic)则是指在不同方向上具有不同的特性。例如,在一个各向同性的数据集中,像素在所有方向上的大小和形状都是相同的,而在各向异性的数据集中,像素在不同方向上可能具有不同的大小和形状。
各向异性的数据集在处理过程中可能会带来一些挑战,因为不同方向上的特性不同,可能需要使用不同的算法或技术来处理数据。例如,在使用图像滤波器或分割算法时,需要考虑各向异性的因素,以确保在所有方向上都能得到准确的结果。
3D U-Net看起来是处理3D图像数据的一个合适的选择。在理想世界中,我们可以在整个患者图像上训练该框架。而在现实中,我们会受到GPU资源的限制,这使得我们只能通过图像patch的方式来训练该框架。基于patch的训练方式对于图像尺寸较小的数据集(比如Brain Tumour数据集,Hippocampus数据集以及Prostate数据集)来说并不是问题,但对于图像较大的数据集(比如Liver数据集),可能会对训练造成阻碍。主要原因在于受限的感受野无法收集足够的上下文信息。
👉U-Net Cascade
为了解决3D U-Net在大尺寸图像上的缺点,我们提出了一个级联模型。首先是stage1,在下采样图像上训练一个3D U-Net。然后是stage2,将这个3D U-Net的分割结果上采样至原始分辨率,并作为额外输入通道喂给下一个3D U-Net,第二个3D U-Net在全分辨率上基于patch进行训练。见Fig1。
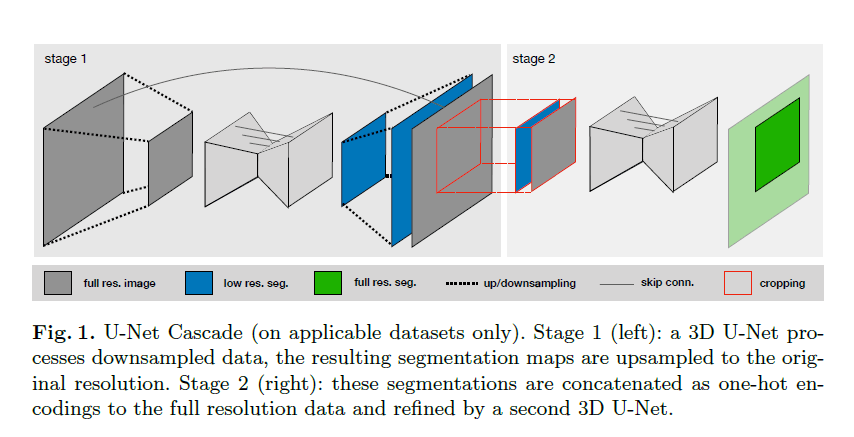
👉Dynamic adaptation of network topologies
由于输入图像的尺寸差异巨大(比如Liver数据集中图像尺寸的中位数为$482 \times 512 \times 512$,而Hippocampus数据集中图像尺寸的中位数为$36 \times 50 \times 35$),必须针对每个数据集自动调整输入的patch大小以及每个方向上pooling操作的次数(从而隐含地调整卷积层的数量),以使得空间信息可以充分聚合。除了自适应图像大小之外,还需考虑内存占用的不同。我们在这方面的原则是动态权衡batch size和网络容量,具体见下:
我们从网络配置开始介绍,这些网络配置需要和硬件相匹配。对于2D U-Net的基本参数配置,我们设置输入patch大小为$256 \times 256$,batch size=42,网络起始卷积层(即最高分辨率层)的feature map数量为30(每次下采样,feature map数量都翻倍)。我们会根据每个数据集图像尺寸分布的中位数来自动调整以上参数。我们会对每个轴都进行pooling操作,直到该轴的feature map维度小于8(但最多不能超过6次pooling操作)。和2D U-Net一样,我们将3D U-Net的最高分辨率层的feature map数量也设置为30。对于3D U-Net的基本参数配置,输入patch大小为$128 \times 128 \times128$,batch size=2。由于内存限制,输入patch的体积不会超过$128^3$个体素,但是patch的长宽比会和数据集中图像尺寸分布的中位数尺寸的比例相匹配。如果数据集的中位数尺寸小于$128^3$,则我们就使用中位数尺寸作为输入patch大小,并增加batch size(使得体素总数和$128 \times 128 \times128$,batch size=2时一致)。和2D U-Net一样,我们最多沿着每个轴做5次pooling,直到feature map的维度小于8。
此外,对于任何网络,我们将每个优化步骤(即每个batch)处理的体素总数(即batch size乘上输入volume的体积)限制在整个数据集体素数量的5%以内。对于超过限制的情况,我们会减小batch size(但最小为2)。
第一阶段(即第1部分提到的开发阶段)所用的所有网络的结构见表1。
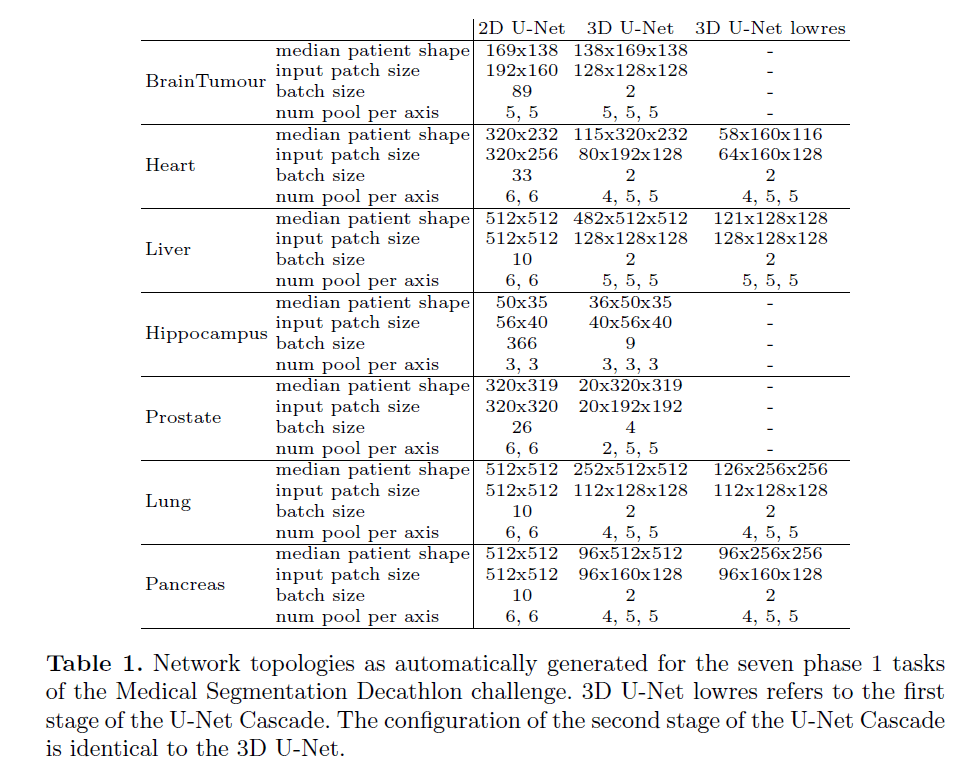
个人理解:在nnU-Net中,一共训练一个2D U-Net和一个3D U-Net,至于是否需要级联U-Net,需要满足一定条件(见下文)。网络的超参数都是根据数据集自适应调整的。
在表1中,3D U-Net lowres指的是级联U-Net中的stage1,3D U-Net指的是级联U-Net中的stage2。
这里结合表1说下nnUnet自动调整参数的方法。
先看Brain Tumour中的2D U-Net,中位数尺寸是$169 \times 138$,按照前文所说方法,我们需要下采样至维度小于8,但如果我们直接把中位数尺寸用作输入patch大小,进行一次下采样后,尺寸就变成了$84.5 \times 69$,这样就产生了小数,为了避免产生小数且满足下采样至维度小于8(或下采样次数满足6次),所以对输入patch大小进行了微调。169通过5次下采样可以使维度小于8,为5.28125,向上取整到6,还原回去(即再进行5次上采样:$6 \times 2^5$)即可得到192。同理,138也是通过5次下采样后维度才小于8,为4.3125,向上取整到5,还原回去为160($5 \times 2^5=160$)。因此,输入patch大小就被微调为$192 \times 160$,并且每个轴向的pooling次数都是5。按照2D U-Net的标准配置,一个batch所包含的像素个数为$256 \times 256 \times 42$,为了保持一个batch内的像素总数基本不变,所以batch size被调整为$256 \times 256 \times 42 \div 192 \div 160 = 89.6$,向下取整为89。其余数据集上的操作类似。
然后再看下Brain Tumour中的3D U-Net,中位数尺寸是$138 \times 169 \times 138$,大于设定的$128^3$,所以输入patch大小直接设为$128 \times 128 \times 128$。每个轴向做5次pooling操作维度才小于8(注意是小于8,不可以等于8)。再看一个Hippocampus数据集的例子,中位数尺寸是$36 \times 50 \times 35$,体素总数小于设定的$128^3$,只需要按照前面的思路微调输入patch大小即可。batch size的计算:$128^3 \times 2 \div 40 \div 56 \div 40 \approx 46.8$,但是这里作者给的是9,应该是要把一个batch处理的体素总数控制在该数据集体素总数的5%。
这里自动调节的参数只有输入patch大小、batch size和pooling次数。
2.2.Preprocessing
预处理步骤也都是自动执行的,不需要用户干预。
👉Cropping
所有数据都只保留非零值区域。这对大部分数据集没有影响,比如liver CT数据集。但是会减少skull stripped brain MRI数据的大小(从而减少计算负担)。
👉Resampling
CNN并不能很好的理解体素间距(voxel spacing)这个概念。在医学图像中,不同的机器或协议通常会产生不同体素间距的数据。为了使我们的网络能够正确的学习空间语义信息,所有数据都被重采样到其所在数据集的体素间距中位数,其中三阶样条插值(third order spline interpolation)用于图像数据,最近邻插值(nearest neighbor interpolation)用于相应的分割掩模。
个人理解:分辨率定义了体素的数量,而体素间距定义了一个体素的物理大小。CT图像中的成像大小就是分辨率乘上体素间距。重采样的目的就是让体素间距变为一致,这样分辨率就可以反映成像大小了。
是否需要使用U-Net级联,可通过以下条件确定:如果重采样后的数据的中位数形状所包含的体素数量是3D U-Net(batch size=2)输入patch所包含体素数量的4倍以上,则可以使用U-Net级联模型,并且数据会被进一步下采样至更低的分辨率。通过将体素间距增加2倍(降低分辨率)来实现下采样,直到刚好满足4倍的关系。如果数据集是各向异性的,则首先对高分辨率轴进行下采样,直到它们与低分辨率轴匹配,然后再对所有轴同时进行下采样。以下数据集满足使用U-Net级联的条件:Heart、Liver、Lung和Pancreas。
👉Normalization
因为CT扫描得到的CT值范围都是固定的,所以相当于CT数据已经进行了一次自动归一化,然后我们对其进行进一步的二次归一化:对于CT数据,统计训练集内分割mask的CT值,取这些CT值在$[0.5,99.5]$百分位数内的值,然后基于这些值的均值和标准差对数据进行z-score归一化。对于MRI和其他类型的数据,则对每个患者数据单独执行z-score归一化。
如果cropping操作将数据集的平均大小(以体素为单位)减少了$\frac{1}{4}$或更多,则归一化只在mask内执行,mask外的值都设为0。
2.3.Training Procedure
所有模型都是从头开始训练的,并在训练集上使用5折交叉验证进行评估。我们使用的损失函数是dice和交叉熵的组合:
\[L_{total}=L_{dice}+L_{CE} \tag{1}\]对于几乎会在所有患者上都运行的3D U-Net(如果使用级联U-Net,则指的是第一阶段的3D U-Net;如果不使用级联U-Net,则指的就是一个单独的3D U-Net),我们会计算batch中每个sample的dice loss,并计算整个batch的平均值。对于其他网络(级联U-Net的第二阶段或2D U-Net),我们会将batch内的所有sample当做一个整体来计算整个batch的dice loss。
我们使用的dice loss见下:
\[L_{dc} = -\frac{2}{\lvert K \rvert} \sum_{k \in K} \frac{\sum_{i \in I} u_i^k v_i^k}{\sum_{i \in I} u_i^k + \sum_{i \in I} v_i^k} \tag{2}\]$u$是softmax的输出,$v$是GT分割结果的one hot编码。$u$和$v$的形状都是$I \times K$,$i \in I$是patch或batch中的像素数,$k \in K$是类别数。
对于所有实验,我们都使用Adam优化器,初始学习率为$3 \times 10^{-4}$。一个epoch包含超过250次迭代。在训练过程中,我们对验证集和训练集的loss都执行指数滑动平均(验证集loss的滑动平均表示为$l_{MA}^v$,训练集loss的滑动平均表示为$l_{MA}^t$)。如果$l_{MA}^t$在连续30个epoch内降低没有超过$5 \times 10^{-3}$,则学习率除以5。如果$l_{MA}^v$在过去连续60个epoch内降低都没有超过$5 \times 10^{-3}$,或学习率小于$10^{-6}$,则训练自动停止。
👉Data Augmentation
当使用有限的数据训练大型神经网络时,必须特别注意防止过度拟合。我们通过各种各样的data augmentation来解决这个问题。我们在训练中使用了以下data augmentation:随机旋转、随机缩放、随机弹性形变、gamma correction、镜像等。data augmentation是用我们自己内部的框架完成的,该框架在github.com/MIC-DKFZ/batchgenerators。
我们对2D U-Net和3D U-Net分别定义了一组data augmentation参数。这些参数在不同数据集之间不会修改。
如果3D U-Net输入patch的最长边是最短边的两倍以上,则应用三维data augmentation可能是次优的。对于该类型的数据集,我们使用2D data augmentation,并对每个sample逐片(slice-wise)使用。
级联U-Net的第二个阶段会接收上一阶段的分割结果作为额外的输入通道。为了防止过于依赖上一阶段的分割结果,我们对其应用了随机的形态学操作(腐蚀、膨胀、开、闭),并随机去除这些分割的连通域(connected components)。
👉Patch Sampling
为了提高网络训练的稳定性,我们强制要求一个batch中超过$\frac{1}{3}$的sample至少包含一个随机的前景类。
2.4.Inference
因为我们的训练都是基于patch的,所以我们的推理也都是基于patch的。由于网络精度在越接近patch边界的地方越低,所以在跨patch聚合预测结果时,我们对靠近中心的体素的权重高于靠近边界的体素。patch的重叠比例为patch size的一半,在推理时使用了镜像方式的data augmentation。
通过组合使用拼接预测(tiled prediction,即重叠的patch)和test time data augmentation(即镜像),一个体素可以有多达64个预测结果。并且,训练时的5折交叉验证也对应产生了5个模型,我们会集成这5个模型的结果来进一步提高鲁棒性。
2.5.Postprocessing
对训练数据的GT分割标签做连通域分析。如果一个类在所有case中都位于一个单独的连通域中,则这种情况可以被解释为数据集的一般属性。因此,在相应数据集的预测图像上,我们也只保留该类的最大连通域。
2.6.Ensembling and Submission
为了进一步提高分割性能和鲁棒性,对于每个数据集,我们使用2D U-Net、3D U-Net和级联U-Net这3个模型分别进行预测,然后将预测结果两两组合。我们会自动选择取得最高平均前景dice分数的模型作为最优模型。
3.Experiments and Results
我们在第一阶段数据集上使用五折交叉验证来优化网络。第一阶段交叉验证结果以及最终提交的测试结果见表2。-表示级联U-Net不适用于该数据集,因为其图像已经完全被3D U-Net的输入patch大小所覆盖。最终提交的模型以粗体显示。尽管平台允许提交多次,但我们认为这是不好的做法。因此,我们就只提交了一次,并报告了这次提交的结果。
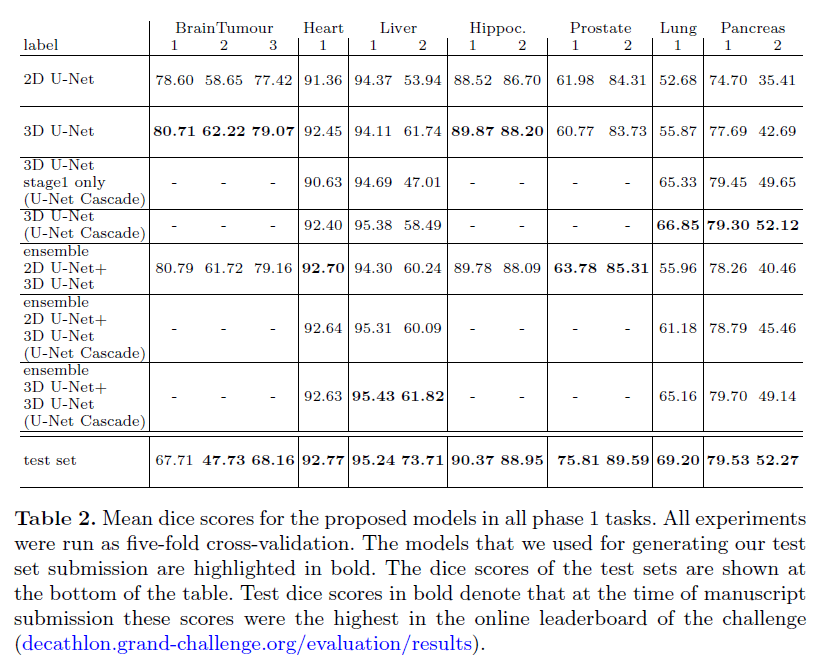
个人理解:说一下表2怎么看:
- 2D U-Net(第一行):仅用2D U-Net,通过五折交叉验证,并对结果求平均,得到在这几个数据集上的dice分数。
- 3D U-Net(第二行):仅用3D U-Net,通过五折交叉验证,并对结果求平均,得到在这几个数据集上的dice分数。可以看到,在验证集上,该模型在BrainTumour和Hippoc.数据集上取得了最优的结果。该模型也被提交用于这两个数据集对应测试集的结果评估(评估结果见test set一行)。
- 3D U-Net stage1 only (U-Net Cascade)(第三行):仅用级联U-Net的stage1,通过五折交叉验证,并对结果求平均,得到在这几个数据集上的dice分数。有些数据集不满足用级联U-Net的要求,所以结果用“-”表示。都是单独的一个3D U-Net,但这一行的结果和第二行不一样,个人认为原因在于第2.2章节的Resampling部分,级联U-Net有不同的前处理方式,会有下采样。
- 3D U-Net (U-Net Cascade)(第四行):使用完整的级联U-Net。在验证集上,该模型在Lung和Pancreas数据集上取得了最优的结果。该模型也被提交用于这两个数据集对应测试集的结果评估(评估结果见test set一行)。
- ensemble 2D U-Net+3D U-Net(第五行):将2D U-Net和3D U-Net集成。在验证集上,该模型在Heart和Prostate数据集上取得了最优的结果。该模型也被提交用于这两个数据集对应测试集的结果评估(评估结果见test set一行)。
- ensemble 2D U-Net+3D U-Net (U-Net Cascade)(第六行):将2D U-Net和级联U-Net集成。
- ensemble 3D U-Net+3D U-Net (U-Net Cascade)(第七行):将3D U-Net和级联U-Net集成。在验证集上,该模型在Liver数据集上取得了最优的结果。该模型也被提交用于这一数据集对应测试集的结果评估(评估结果见test set一行)。
- test set(第八行):测试集上的结果。粗体表示优于所有的竞争对手,排名第一。
从表2中可以看出,通过交叉验证得到的结果比较稳健,在测试集上没有出现过拟合。唯一一个性能有明显下降的是BrainTumour数据集。原因是验证集和测试集的数据分布有着巨大不同。
4.Discussion
在本文中,我们提出了用于医学领域的nnU-Net分割框架,该框架直接围绕原始的U-Net框架进行构建,并动态适应任何给定的数据集。基于我们的假设,非框架性质的修改比框架修改要强大得多。适应新分割任务所需的所有设计选择都是以完全自动的方式完成的,无需手动交互。对于每个任务,nnU-Net会自动对三种不同且自动配置的U-Net进行五折交叉验证,并选择具有最高平均前景dice分数的模型(或集成模型)作为最终提交。在医学分割十项全能挑战中,我们证明了nnU-Net在7个高度不同的医学数据集的测试集上表现得都很有竞争力,在我们提交时,在排行榜上所有任务的所有类别都取得了最高的dice分数(除了BrainTumour的类别1)。我们承认,训练三个模型并为每个数据集独立选择最好的模型并不是最简洁的解决方案。如果有更多时间,可以在训练之前研究适当的启发式方法来确定给定数据集的最佳模型。我们目前的想法是倾向于级联U-Net(如果不满足使用级联U-Net,则使用3D U-Net),唯一的例外是Prostate任务和Liver任务。此外,我们许多设计的额外好处没有得到适当的验证,比如使用Leaky ReLU代替传统的ReLU以及我们data augmentation的参数设置。在未来的工作中,我们会侧重于通过消融实验来系统性的评估所有这些设计选择。
5.原文链接
👽nnU-Net:Self-adapting Framework for U-Net-Based Medical Image Segmentation