本文为原创文章,未经本人允许,禁止转载。转载请注明出处。
1.Introduction
在医学图像处理中,我们希望的是localization,即每个像素点的类别标签。此外,在医学图像领域,数据量通常不多。
本文我们基于FCN提出一种更简洁的网络结构(见Fig1),其只需要少量的训练数据就可产生不错的分割结果。
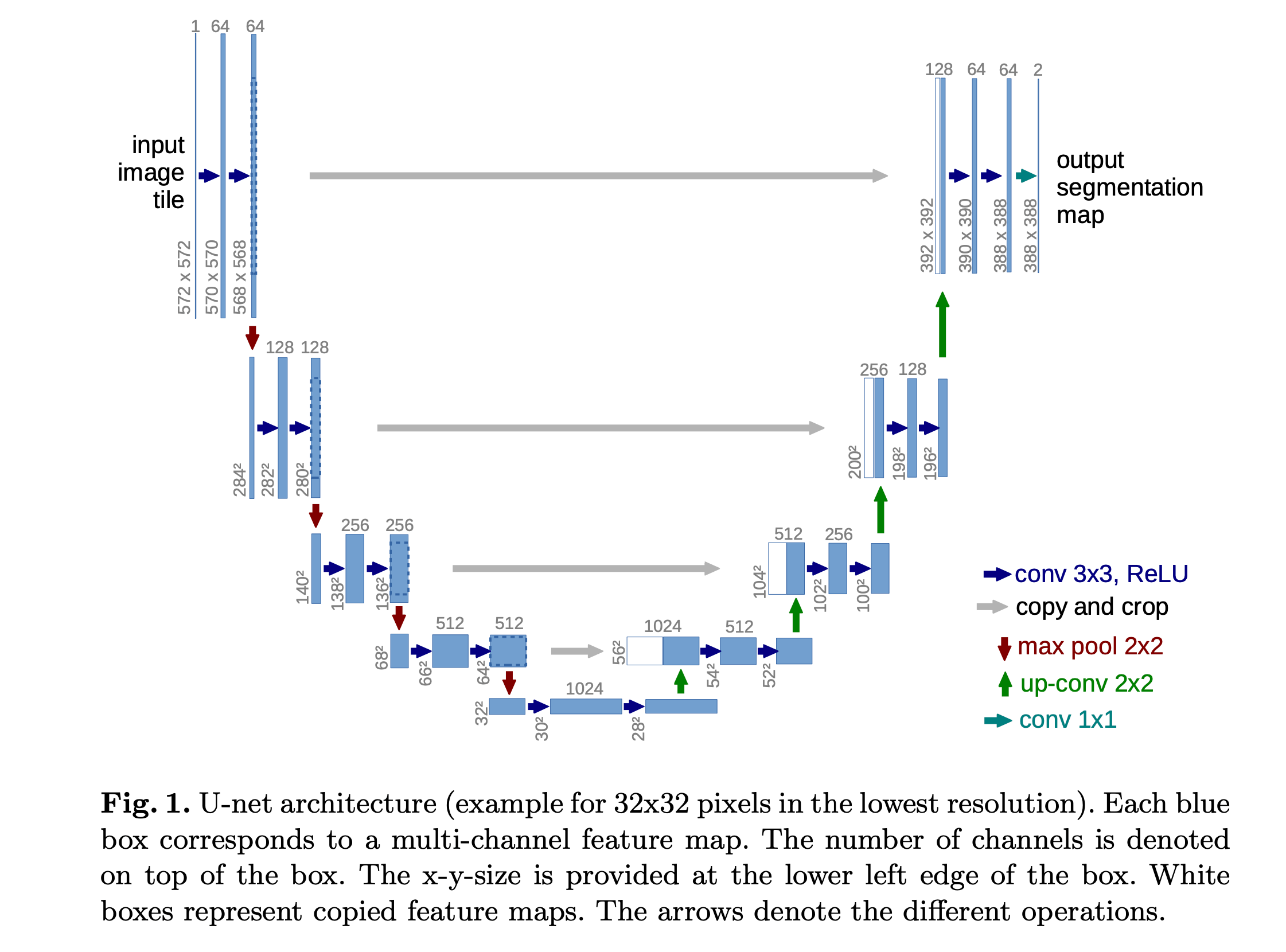
Fig1中,每个蓝色块代表一个多通道的feature map,通道的数量标注在蓝色块的上方。feature map的x,y大小标注在蓝色块的左侧。白色块表示是拷贝过来的feature map。不同颜色的箭头代表不同的操作。
相比FCN,U-Net的一个重要修改是在上采样时,增加了feature map的通道数,方便网络将信息更好的传递到高分辨率层。U-Net呈U型结构,网络左侧的contracting path和右侧的expansive path基本是对称的。U-Net没有使用全连接层,padding方式均为VALID。U-Net可以通过overlap-tile策略对任意大小的图像进行无空隙的分割(见Fig2)。
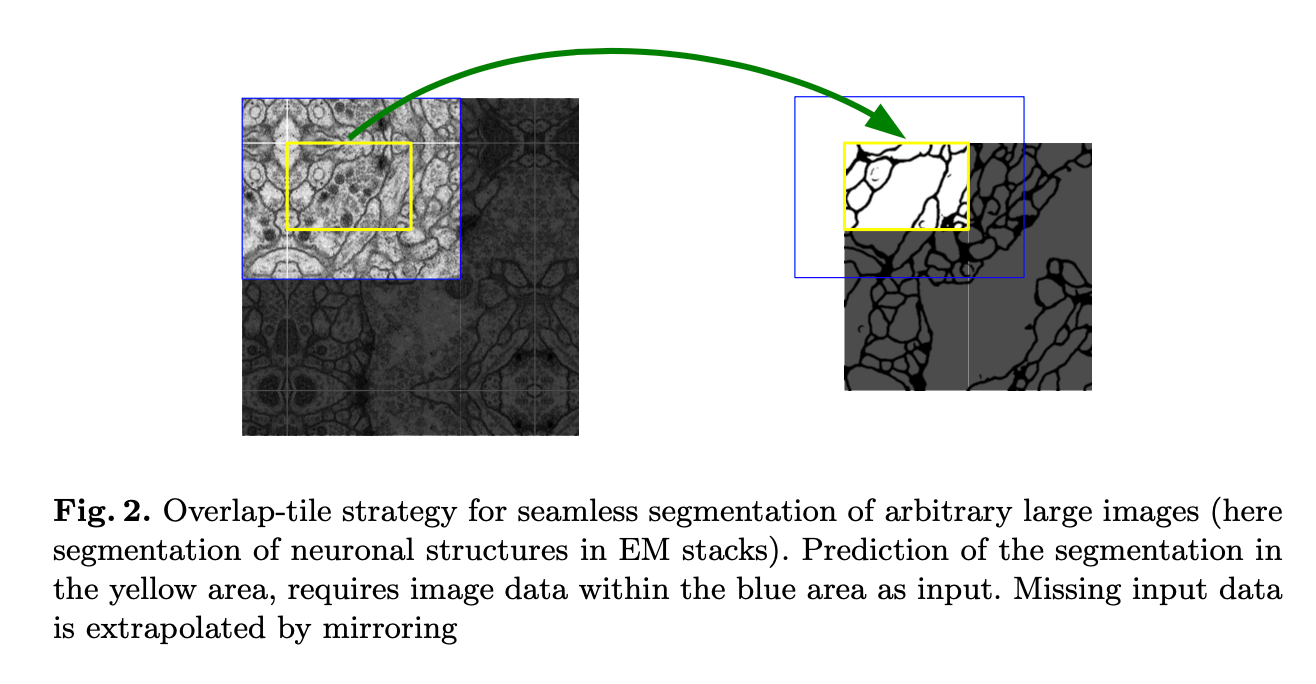
通过U-Net的结构可以发现,其输出的维度是小于输入维度的。因此在Fig2中,蓝色框是输入的大小,黄色框是输出的结果。如果蓝色框里的数据不完整,则通过镜像的方式补全。这种tiling的策略对于将U-Net应用于大型图像非常重要,否则容易受到GPU内存的限制。
因为我们的任务中训练数据非常少,所以我们使用弹性形变(elastic deformations)来进行数据扩展。这使得网络可以学习到不同形变中的共通性。这在医学图像分割中尤其重要,因为形变是细胞组织中最常见的变化,可以有效模拟真实情况。
对于许多细胞分割任务来说,另一个挑战是区分互相接触且属于同一类别的目标(见Fig3)。因此,我们建议使用加权loss,将互相接触的细胞分割开来的区域(即被预测为背景)应该赋予更大的loss权重。

由此产生的网络适用于各种医学图像分割问题。
2.Network Architecture
网络结构见Fig1。最后通过$1\times 1$卷积将64通道降为2通道,因为一共有两个类别。该网络一共有23个卷积层。
3.Training
输入图像及其对应的segmentation maps被用于训练网络,使用Caffe框架。因为卷积层都没有使用padding,所以输出的尺寸比输入要小(差值是一个恒定的值)。为了最小化开销并最大化GPU利用率,我们倾向于使用较大的input tiles,而不是大的batch size,因此我们将batch size设为单张图像(个人理解:batch size=1)。设momentum=0.99。
能量函数是基于最终的feature map的像素级别的softmax函数和交叉熵损失函数的结合。softmax函数的定义为:
\[p_k(\mathbf{x}) = exp( a_k(\mathbf{x}) ) / ( \sum^K_{k'=1} exp (a_{k'}(\mathbf{x})) )\]$a_k(\mathbf{x})$表示第$k$个feature map在某一像素坐标的激活值。feature map的通道数$K$就是类别个数,每个通道代表一个类别。经过softmax函数之后,同一像素坐标但不同通道的点只能归属于概率最大的那个类别。交叉熵损失函数定义为:
\[E=\sum_{\mathbf{x} \in \Omega} w(\mathbf{x}) \log (p_{\ell (\mathbf{x})} (\mathbf{x})) \tag{1}\]$p_{\mathcal{l}(\mathbf{x})} (\mathbf{x})$为$\mathbf{x}$归属于真实类别$\ell$的概率。$w$为权重。
权重是根据GT预先计算好的,添加权重是为了让网络可以学习到小的分割边界,比如相互接触的细胞之间的边界(见Fig3中的c和d),这类边界点的权重很高。
权重的计算见下式:
\[w(\mathbf{x}) = w_c (\mathbf{x}) + w_0 \cdot exp \left( -\frac{(d_1(\mathbf{x}) + d_2(\mathbf{x}))^2}{2\sigma^2} \right) \tag{2}\]$w_c$是一个weight map,用于解决类别不平衡。$d_1$是到最近的细胞的边界的距离,$d_2$是到第二近的细胞的边界的距离。根据我们的经验:$w_0=10,\sigma \approx 5$。
网络的权值初始化非常重要,我们使用高斯分布对权重进行初始化。高斯分布的标准差为$\sqrt{2/N}$,$N$表示一个神经元的传入节点数。例如,如果卷积核大小为$3\times 3$,数量为64,则$N=9\cdot 64=576$。
3.1.Data Augmentation
当训练样本过少时,数据扩展是增加网络鲁棒性的一个重要方法。对于显微图像,我们主要需要平移和旋转不变性,以及对变形和灰度变化的鲁棒性。我们所使用的弹性形变是这类数据扩展的一个重要方式。针对弹性形变,我们使用$3\times 3$的高斯核,且$\sigma = 10$,插值方式采用bicubic interpolation。
4.Experiments
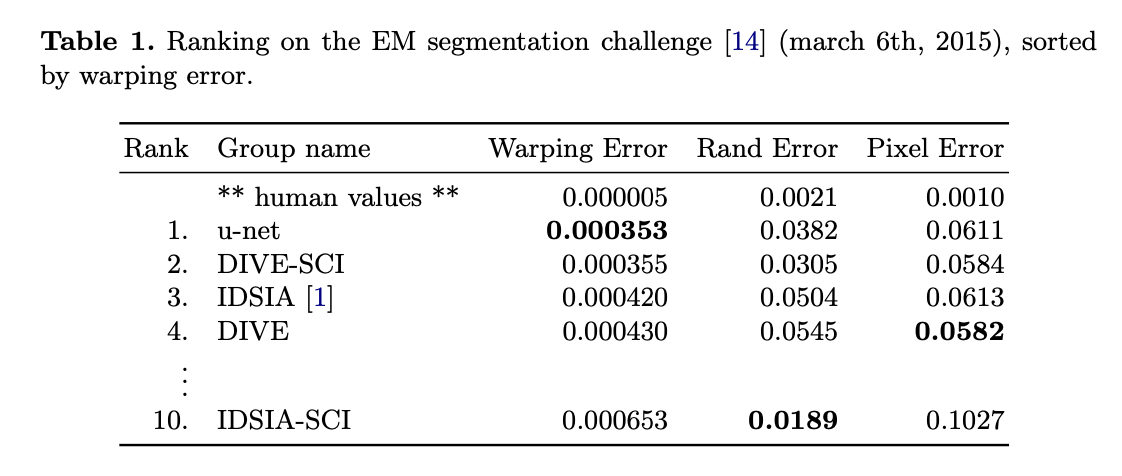
我们在三个不同的分割任务上测试了u-net。第一个任务是分割电子显微镜下的神经元结构。数据集示例以及我们的分割结果见Fig2。训练集有30张图像($512\times 512$)。训练集中每幅图像都有GT。测试集误差从三个方面评估,分别是:warping error、rand error和pixel error,比较结果见表1:
第二个和第三个任务是细胞分割。第二个任务使用“PhC-U373”数据集,训练集包含35张部分标记的图像,u-net的结果见Fig4的a列和b列,平均IoU达到了92%。第三个任务使用“DIC-HeLa”数据集(见Fig3和Fig4c),训练集包含20张部分标记的图像,我们方法的平均IoU达到了77.5%。

5.Conclusion
u-net在生物医学分割任务中表现很好。通过弹性形变进行数据扩展使得我们仅需少量的标记数据便可以达到不错的结果。u-net的官方实现以及相关材料见:http://lmb.informatik.uni-freiburg.de/people/ronneber/u-net。
6.原文链接
👽U-Net: Convolutional Networks for Biomedical Image Segmentation